9月21日,中國科學(xué)院深圳先進技術(shù)研究院劉陳立課題組與傅雄飛課題組合作在Molecular Systems Biology上發(fā)表了題為“Exploiting spatial dimensions to enable parallelized continuous directed evolution”的文章。該項工作定量研究了細(xì)菌-噬菌體在空間上共同生長遷移的動力學(xué)過程,并基于這一定量理解,發(fā)展了空間噬菌體輔助連續(xù)定向進化系統(tǒng)(Spatial Phage-Assisted Continuous Evolution, SPACE),實現(xiàn)了合成生物元件的大規(guī)模平行進化。
深圳先進院合成所劉陳立研究員、傅雄飛研究員為該文章的通訊作者,助理研究員魏婷、賴旺生、博士研究生陳茜、及助理研究員張易為該文章的共同第一作者。

文章上線截圖
文章鏈接:https://doi.org/10.15252/msb.202210934
合成生物學(xué)旨在以工程化的理念改造或創(chuàng)造人工生物系統(tǒng),以期理解生命、改造生命、合成生命乃至設(shè)計生命。生物元件作為合成生物系統(tǒng)的基本單元,它的數(shù)量和功能制約著合成生物學(xué)的發(fā)展。傳統(tǒng)手段通過挖掘天然生物系統(tǒng)獲得的生物元件,其性能常常不能滿足人們的需求;通過理性設(shè)計發(fā)展全新生物元件的方式雖有潛力,但目前還受限于設(shè)計能力;而通過定向進化,利用進化這個“上帝之手”的力量來對已有元件進行改造和優(yōu)化獲得所需的功能,已成為合成生物學(xué)領(lǐng)域中一項重要的關(guān)鍵平臺性技術(shù)。
傳統(tǒng)的定向進化方法一般分為突變建庫和篩選這兩個步驟[1],循環(huán)迭代需要大量的重復(fù)操作,耗費人力物力和時間。連續(xù)定向進化方法(continuous directed evolution)則利用可自我復(fù)制的生物體,在其基因組復(fù)制過程引入突變、并利用突變后該生物體復(fù)制擴增能力的差異性變化來實現(xiàn)建庫與篩選這兩個步驟的自動連接和迭代循環(huán),從而減少人力勞動,使定向進化快速進行[2]。2011年,美國哈佛大學(xué)David Liu實驗室開發(fā)了噬菌體輔助連續(xù)進化(Phage-Assisted Continuous Evolution, PACE)[3],將M13噬菌體的增殖與待進化的生物分子功能關(guān)聯(lián)在一起,在一天內(nèi)就實現(xiàn)了數(shù)十輪的定向進化,大大提升了進化效率,是定向進化技術(shù)發(fā)展中里程碑式的重要突破。PACE已被應(yīng)用于RNA聚合酶、TALEN、Cas9、堿基編輯器等重要酶類的進化[3-6]。
然而,如果我們要同時進化多個目標(biāo)蛋白,目前還缺少一種簡便的定向進化技術(shù)/方法;另一方面,PACE系統(tǒng)需要連續(xù)培養(yǎng)裝置(恒化器,chemostat)、復(fù)雜的流速控制與檢測設(shè)備和一定的操作技巧,因此普通實驗室不太容易開展PACE實驗。新發(fā)展的SPACE系統(tǒng)將復(fù)雜的進化裝置簡化成一個每個生物實驗室都有的瓊脂平板,很好的解決了這兩個難題。
定量解析宿主-病毒共遷移系統(tǒng)的進化時空動力學(xué)
SPACE系統(tǒng)的開發(fā)靈感來源于劉陳立課題組前期關(guān)于細(xì)菌遷移定植的研究(詳見:專家點評Nature | 劉陳立/華泰立合作利用遷徙進化實驗揭示合成生物建構(gòu)原理)[7]。在前期研究中,團隊發(fā)現(xiàn)處在空間擴張前鋒(front)的細(xì)菌始終保持在對數(shù)生長期,而落后于前鋒的細(xì)菌不再進行定向遷移運動并繼續(xù)生長進入平臺期。這不就是一個“移動的恒化器”嗎(圖1A)?
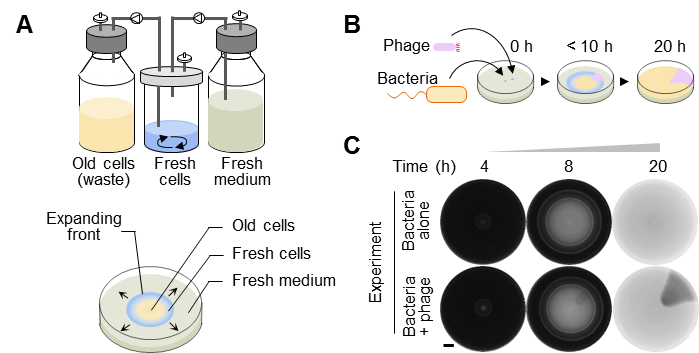
圖1 細(xì)菌空間擴展過程中的噬菌體感染
A,細(xì)菌空間擴展系統(tǒng)與連續(xù)培養(yǎng)裝置的相似性;B,細(xì)菌-噬菌體共遷移實驗示意圖;C,實驗結(jié)果
本項目研究團隊原創(chuàng)性地利用這個“移動的恒化器”替代PACE系統(tǒng)中噬菌體的持續(xù)擴增所需要的連續(xù)培養(yǎng)裝置,從而大幅簡化了系統(tǒng)。研究團隊構(gòu)建了細(xì)菌-噬菌體共遷移實驗體系,使得原本沒有運動能力的噬菌體被處于空間擴張運動過程中的宿主細(xì)菌攜帶并廣泛傳播。由于被感染的細(xì)菌生長速率慢于未被感染的細(xì)菌,最終在軟瓊脂表面的菌苔上產(chǎn)生肉眼可見的低細(xì)菌密度的扇形感染區(qū)(圖1BC)。雖然宿主-病毒共遷移系統(tǒng)在自然界普遍存在,但人們對遷移過程中病毒如何進化了解的很少。
為了理解這一過程,劉陳立課題組與傅雄飛課題組合作,以經(jīng)典的傳染病模型SIR(易感者Susceptible,感染者Infected,康復(fù)者Recovered)為基礎(chǔ),結(jié)合細(xì)菌空間擴張運動模型,建立了空間擴張系統(tǒng)的宿主感染模型(Range Expansion with Susceptible Infected Recovered kinetics)(圖2)。該數(shù)學(xué)模型刻畫了空間擴張體系下細(xì)菌-噬菌體共遷移的時空動力學(xué),解釋了扇形噬菌體感染區(qū)的形成過程(圖1C)。結(jié)合模型模擬與定量實驗,研究團隊發(fā)現(xiàn)扇形感染區(qū)的面積與噬菌體感染并產(chǎn)生子代的能力呈正相關(guān)關(guān)系,這使噬菌體感染能力的強弱有了直觀的可視化表征方式。
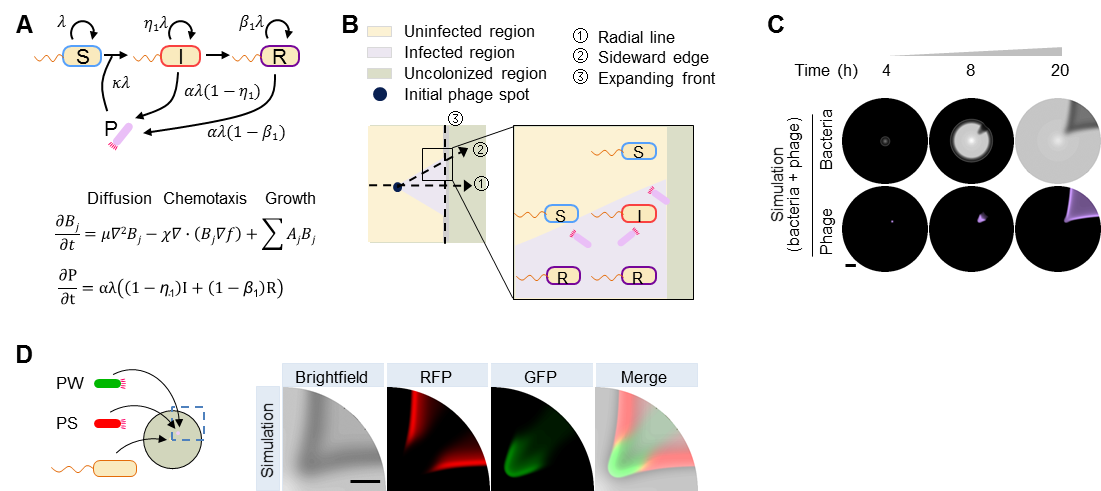
圖2 空間擴展過程中噬菌體與細(xì)菌互作動力學(xué)模型
A,細(xì)菌SIR三種狀態(tài)的轉(zhuǎn)化及噬菌體生產(chǎn)的數(shù)學(xué)描述;B,感染動態(tài)在不同位置和方向上的差異性;C,模型模擬噬菌體感染區(qū)的形成動態(tài);D,模型模擬強弱噬菌體初始比例為PS:PW=1:105 的空間競爭
隨后,研究團隊利用理論模型結(jié)合實驗進化的方式,解析細(xì)菌遷移過程中噬菌體感染宿主的進化動力學(xué)。對于宿主而言,它們的進化主要發(fā)生在擴展運動的前鋒中,這種現(xiàn)象被稱為空間分選進化(spatial sorting)[8]。研究團隊一開始認(rèn)為病毒的進化也主要發(fā)生在同樣的位置。然而,后續(xù)研究中團隊觀察到了與該預(yù)期截然不同的結(jié)果:與更高的感染能力相關(guān)的噬菌體有益突變快速積累的位置并不是在宿主運動的前鋒中,而是沿著扇形感染區(qū)域的側(cè)邊沿。該現(xiàn)象產(chǎn)生的原因與側(cè)邊沿處存在的非平衡的感染狀態(tài)有關(guān)。這一發(fā)現(xiàn)可能也適用于其它生態(tài)系統(tǒng)的宿主/病毒共遷徙現(xiàn)象。
理性設(shè)計構(gòu)建空間連續(xù)定向進化系統(tǒng)
基于上述定量理解,團隊設(shè)計構(gòu)建了SPACE系統(tǒng)(圖3)。為了適應(yīng)SPACE在半固體培養(yǎng)基中的應(yīng)用,對宿主菌攜帶的一個關(guān)鍵誘變質(zhì)粒進行了功能改造,使具有誘變效果的基因只在宿主細(xì)菌被噬菌體感染之后表達(dá)。未進化的初始噬菌體感染能力很弱,幾乎不能在宿主細(xì)菌的空間擴展過程中形成可見的扇形感染區(qū)。如果誘變過程中其攜帶的待進化基因產(chǎn)生了有益突變,則可以產(chǎn)生感染能力增強的子代噬菌體,從而使其感染區(qū)域面積顯著擴大。
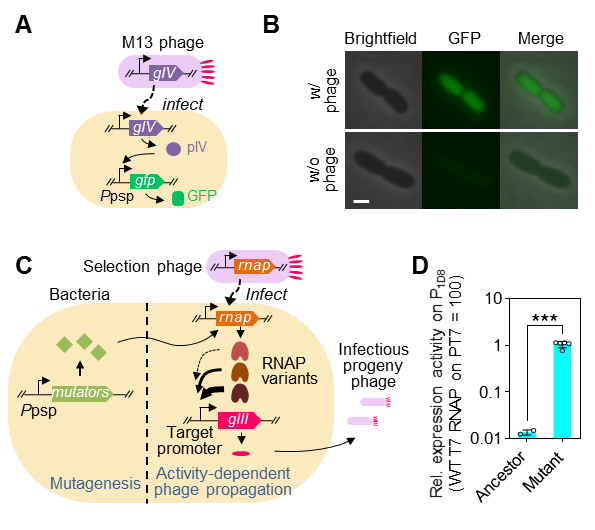
圖3 SPACE系統(tǒng)的構(gòu)建
A, 利用噬菌體休克蛋白(phage shock protein, psp)啟動子改造誘變模塊;B, psp啟動子僅在存在噬菌體感染的條件下啟動表達(dá);C,SPACE系統(tǒng)基因線路設(shè)計;D,T7 RNA聚合酶進化前后識別目標(biāo)啟動子的活性變化
SPACE實現(xiàn)大規(guī)模平行定向進化
SPACE系統(tǒng)具有可視化、簡便的優(yōu)勢,可以實現(xiàn)大規(guī)模平行進化實驗。研究團隊利用該系統(tǒng)平行進化了T7 RNA聚合酶識別隨機啟動子序列的能力(圖4),獲得了一系列啟動子變體-RNA聚合酶突變庫,可用于進一步改造及工程應(yīng)用。
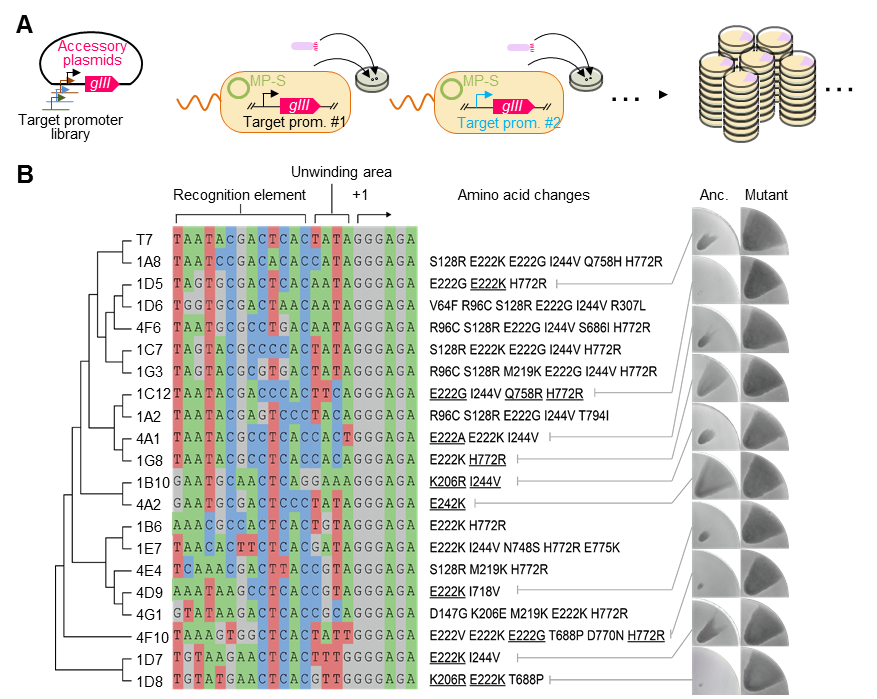
圖4 利用SPACE進行大量平行進化
A,平行進化T7 RNA聚合酶對人工啟動子庫識別活性的實驗示意圖;B,進化獲得的能夠識別目標(biāo)啟動子的聚合酶突變體
本項工作著眼于合成生物學(xué)研究中可用元件匱乏這一亟待解決的關(guān)鍵問題,在現(xiàn)有連續(xù)定向進化方法的基礎(chǔ)上,通過實驗與模型模擬相結(jié)合的方式定量研究了宿主細(xì)菌的空間遷移運動對其噬菌體進化的作用規(guī)律,并由此利用空間維度發(fā)展了蛋白質(zhì)等生物分子的連續(xù)定向進化方法,示范了定量合成生物學(xué)的研究范式。SPACE系統(tǒng)可以在普通實驗室中實現(xiàn)生物元件的大規(guī)模平行進化改造,為合成生物學(xué)在化工、醫(yī)療等領(lǐng)域的應(yīng)用提供豐富的元件庫。同時,細(xì)菌空間遷移運動對噬菌體進化的作用規(guī)律,也將對于研究更高等的生物如昆蟲、鳥類的遷徙對其攜帶的可能對人類健康也存在影響的病毒如何進化等生態(tài)學(xué)理論或調(diào)查研究提供參考和示范。
該項工作獲得了中科院先導(dǎo)專項B、科技部重點研發(fā)計劃、國家自然科學(xué)基金、中科院交叉學(xué)科創(chuàng)新團隊等項目支持。
[1] Bloom JD, Arnold FH. In the light of directed evolution: Pathways of adaptive protein evolution [J]. Proceedings of the National Academy of Sciences, 2009, 106(supplement_1): 9995-10000.
[2] Badran AH, Liu DR. In vivo continuous directed evolution [J]. Curr Opin Chem Biol, 2015, 24(1-10.
[3] Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules [J]. Nature, 2011, 472(7344): 499-503.
[4] Hubbard BP, Badran AH, Zuris JA, Guilinger JP, Davis KM, Chen L, Tsai SQ, Sander JD, Joung JK, Liu DR. Continuous directed evolution of DNA-binding proteins to improve TALEN specificity [J]. Nat Methods, 2015, 12(10): 939-942.
[5] Miller SM, Wang T, Randolph PB, Arbab M, Shen MW, Huang TP, Matuszek Z, Newby GA, Rees HA, Liu DR. Continuous evolution of SpCas9 variants compatible with non-G PAMs [J]. Nat Biotechnol, 2020, 38(4): 471-481.
[6] Thuronyi BW, Koblan LW, Levy JM, Yeh WH, Zheng C, Newby GA, Wilson C, Bhaumik M, Shubina-Oleinik O, Holt JR, Liu DR. Continuous evolution of base editors with expanded target compatibility and improved activity [J]. Nat Biotechnol, 2019, 37(9): 1070-1079.
[7] Liu W, Cremer J, Li D, Hwa T, Liu C. An evolutionarily stable strategy to colonize spatially extended habitats [J]. Nature, 2019, 575(7784): 664-668.
[8] Shine R, Brown GP, Phillips BL. An evolutionary process that assembles phenotypes through space rather than through time [J]. Proceedings of the National Academy of Sciences, 2011, 108(14): 5708-5711.
附件下載:


