腸道菌群的多樣性深刻影響著人類健康,其失調已被證明與糖尿病、阿爾茨海默癥等多種代謝性疾病密切相關。然而,腸道微生物的復雜性與個體差異性給傳統研究方法帶來了巨大挑戰,使得精準解讀和調控腸道微生物成為科學研究的前沿課題。近年來,基因組規模代謝模型(Genome-scale metabolic model,GEM)作為一種包含基因組、生化和生理數據的數學工具,為預測微生物代謝活性提供了可能性。然而,單一GEM僅能對單一菌株的代謝能力進行表征。為了更全面和系統地研究微生物群落,通過整合多種單一菌株模型和宏基因組數據構建群落水平模型(Community-level GEM,Co-GEM),可以預測不同菌株之間的相互作用及群落整體代謝能力,揭示不同物種在群落中的功能角色以及與宿主疾病之間的潛在機制,并在指導設計合成微生物群落實現精準醫療和個性化飲食等領域展現了廣闊的應用潛力。
近日,中國科學院深圳先進技術研究院陳禹課題組與查爾姆斯理工大學Jens Nielsen教授合作,在Current Opinion in Biotechnology期刊發表綜述文章“Personalized gut microbial community modeling by leveraging genome-scale metabolic models and metagenomics”。陳禹研究員和Jens Nielsen教授為文章的共同通訊作者,研究助理李龍濤為第一作者。該工作獲得了國家重點研發計劃及深圳合成生物學創新研究院的支持。
文章首先回顧了近些年GEM相關資源與建模工具(如AGORA2,CarveMe等)及其在腸道微生物研究中的應用,然后介紹了構建個性化人類腸道Co-GEM的兩種主流策略 (圖1):一是通過宏基因組中獲得的微生物分類信息與已有的多個菌株的GEM資源整合構建Co-GEM;二是直接利用宏基因組測序數據構建GEM并結合環境中微生物分類信息來構建Co-GEM。
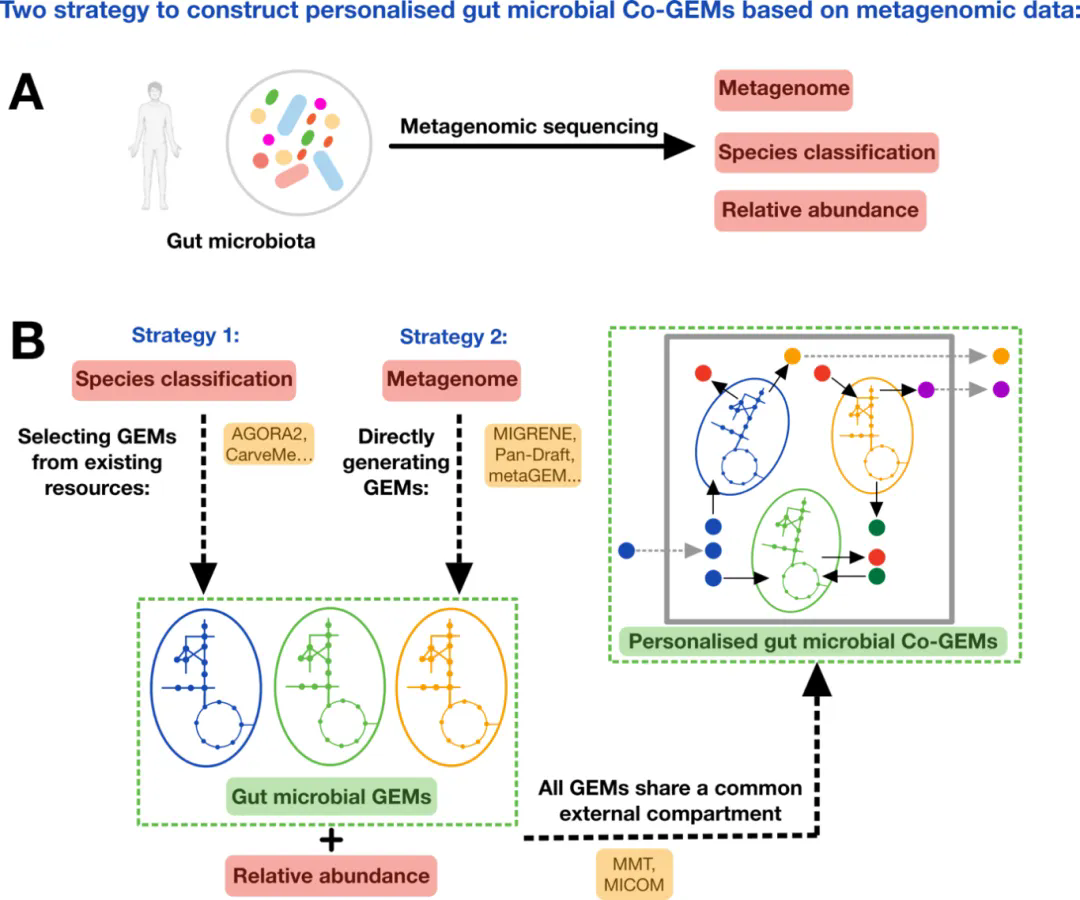
基于宏基因組數據構建個性化腸道微生物Co-GEM的策略
最后,文章總結了該領域的挑戰與展望。首要挑戰便是不同數據庫與GEM資源之間的標準化,目前不同GEM和數據庫之間的代謝物、反應等關鍵信息存在多種不同的格式和命名規則。單一模型的性能是群落建模的基礎,基于先驗知識對GEM進行多約束(比如酶動力學參數,蛋白限制等)的整合至關重要。例如,最新的GECKO 3.0工具通過構建酶約束模型顯著提升了模型的預測能力,有望運用于腸道微生物模型構建。此外,新的“泛”模型構建方法,比如MIGRENE和Pan-draft等,使得構建個性化腸道Co-GEM成為了可能;而多組學數據的整合以及機器學習和神經網絡方法也能夠進一步提升模型性能。隨著新方法的不斷涌現并應用在提高Co-GEM的性能上,相信在不久的將來,將能從腸道微生物的角度為人類健康與疾病提供更深入的見解。
PI和課題組簡介
陳禹,中國科學院深圳先進技術研究院研究員,博士生導師。近五年代表性成果發表于Nature Protocols、PNAS、Molecular Systems Biology等期刊。目前主持國家高層次人才項目、國家重點研發計劃“合成生物學”重點專項和深圳市醫學研究專項資金項目。陳禹課題組從事系統生物學和合成生物學研究,致力于整合“干”“濕”技術定量解析代謝調控機制,提高生物系統理性設計能力。
附件下載:


