本研究依托深圳合成生物研究重大科技基礎設施,開發了一種自動化蛋白質工程方法BO-EVO, 通過多輪機器學習與機器實驗迭代,大幅提升了蛋白質多位點組合突變設計空間的探索效率,能夠以<1%濕實驗量尋找獲得全局最優實驗結果。除利用文獻數據、理論模型數據進行驗證外,BO-EVO方法被應用于實際蛋白質工程任務,4周內將鼠李糖脂合成酶RhlA的酶底物特異性提升了4.8倍。

文章上線截圖
文章鏈接:https://doi.org/10.1093/bib/bbac570
蛋白質適應度地形(fitness landscape)隱喻蛋白質氨基酸序列與其目標性質(“適應度”)對應關系形成的高維表面。蛋白質工程改造可視為在這個高維表面上尋找高點對應的序列。然而有效探索該地形將面臨幾個挑戰:1. 探索空間隨序列長度指數增長;2. 有功能的蛋白極其稀少且高性能蛋白數量隨其適應度呈指數衰減;3. 由于序列位點間突變存在上位效應,地形極度崎嶇;4. 實驗表征費時、費力、費錢。定向進化采用多輪的隨機突變與高通量篩選對適應度地形開展探索,通常每輪固定一個最佳突變。然而,由于適應度地形通常較為崎嶇,貪婪策略更容易陷入局部最優。
在該研究中,機器學習在建模蛋白質適應度地形及指導蛋白質工程方面都取得了初步成功。然而,一方面受限于序列特征不充分及適應度標簽數據稀疏,機器學習模型性能有限;另一方面受限于人力實驗,現有機器學習指導的蛋白工程改造都追求盡可能小的實驗通量和盡可能少的迭代輪次(甚至無迭代)。生物鑄造廠(Biofoundry)通過物理與信息的自動化可以加速生物工程的“設計-構建-測試-學習”閉環過程。在蛋白質工程任務上,自動化、高通量實驗平臺使得短時間內大批量建庫和篩選成為可能,從而能夠獲取高通量、高質量的序列-功能關系數據,用于機器學習模型性能及序列設計質量的閉環迭代提升。
為了實現機器學習算法與機器人實驗之間的高效反饋,數據獲取、模型構建及序列推薦三者缺一不可,需形成閉環。就算法而言,貝葉斯優化正適用于這種復雜適應度地形的尋優,其可利用預測模型的不確定性構建采樣函數以平衡“探索與利用”。然而,經典的貝葉斯優化方法并不適用于高維問題及批量采樣。本研究針對蛋白質多位點組合突變設計空間的高維搜索難題,開發了貝葉斯優化指導的進化算法(BO-EVO,圖1)。作者在研究中提出了搜索空間演化策略,克服經典貝葉斯優化的可擴展性問題,采用迭代式批量化采樣策略,實現與高通量、自動化實驗平臺的高效適配。
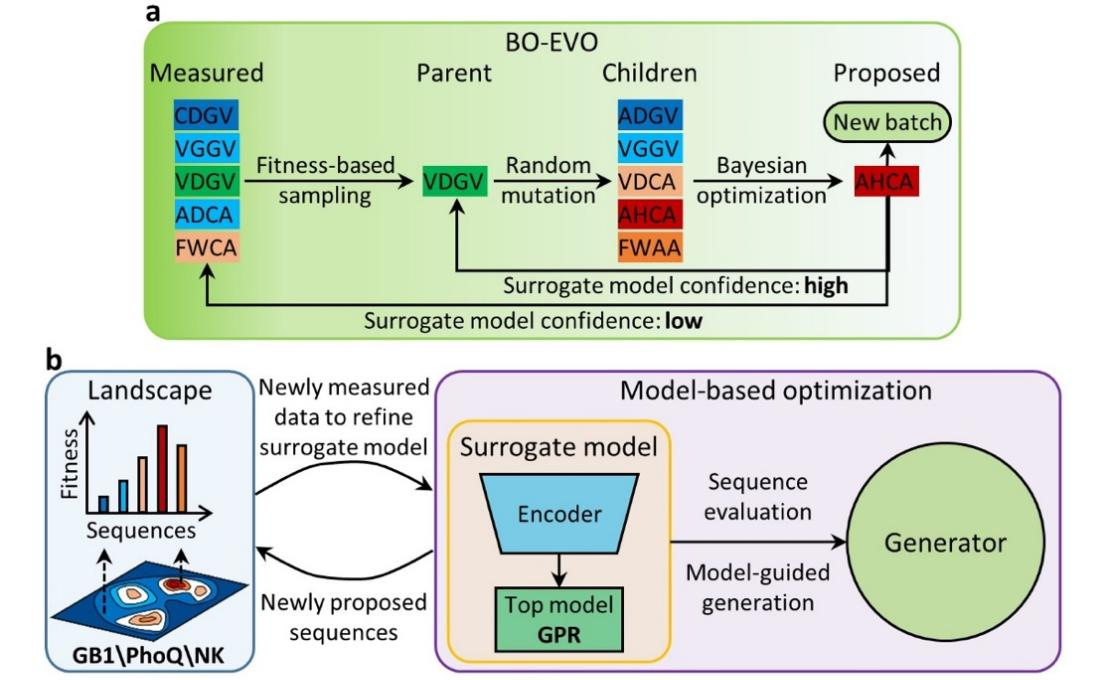
圖1. BO-EVO原理圖。a. BO-EVO算法流程展示搜索空間演化; b. FAST-HIT軟件框架實現數據獲取、模型構建與序列推薦的閉環迭代。
文章以四位點組合突變的GB1經驗地形數據開發算法,確定算法超參。在GB1地形上,BO-EVO性能超越隨機算法、純進化算法(AdaLead)甚至MCMC算法(圖2);其性能雖不及全空間枚舉的貝葉斯優化方法,但計算效率大大提高,可擴展性極好。
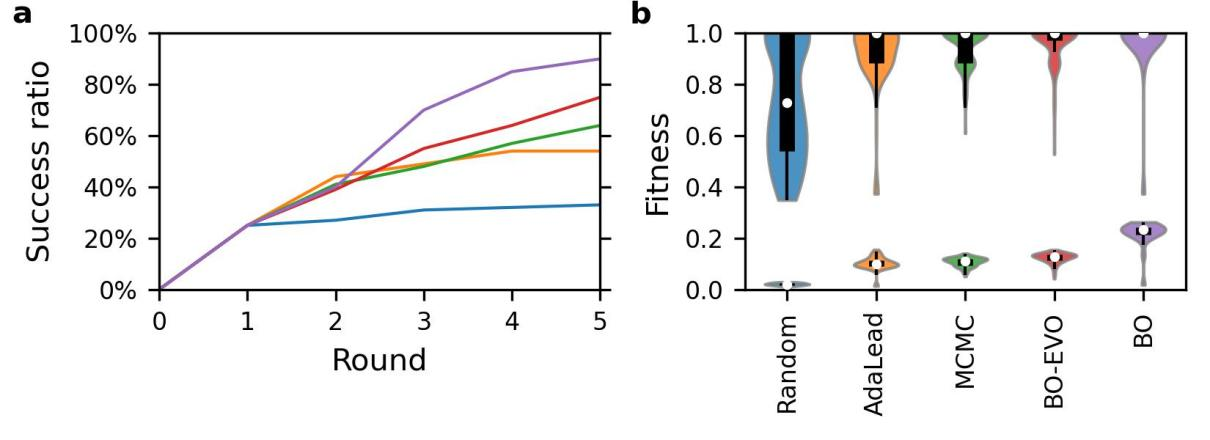
圖2. 適應度地形探索算法比較。a. 找到全局最優的成功率;b. 算法所推薦的序列的適應度均值及最大值。
為了考察對不同適應度地形的適用性,作者將開發好的BO-EVO算法(超參不變)應用在另外一個四位點組合突變的經驗地形PhoQ上,獲得了更高的成功率(圖3)。此外,作者進一步挑戰BO-EVO,將其用在具有不同粗糙度的NK模擬地形(統計模型,粗糙度可控)上,發現對于中等及以下粗糙度的NK地形,BO-EVO表現優秀,而對極其崎嶇的地形,BO-EVO的成功率明顯下降(圖3)。當然,對于極其崎嶇的地形,其適應度的可預測性本身極大降低。
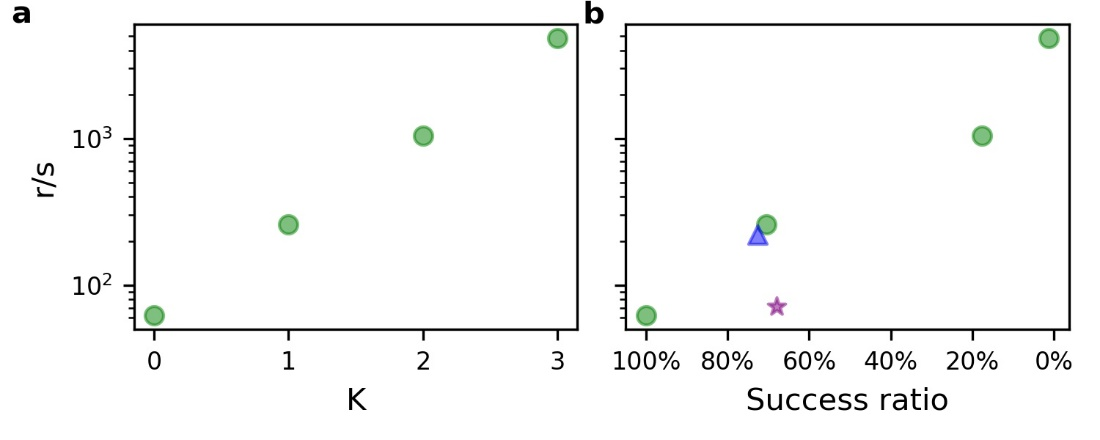
圖3. BO-EVO在不同地形上的泛化能力。a. NK地形的粗糙度;b. BO-EVO迭代5輪后找到全局最優的成功率。綠色圓圈代表NK地形,藍色三角形代表PhoQ地形,紫色五角星代表GB1地形。
最后,作者將BO-EVO算法成功應用于真實的蛋白質工程任務上。面向4位點組合突變的優化任務,作者以鼠李糖脂合成酶RhlA為研究對象,基于BO-EVO指導機器人平臺開展自動化實驗,進行共4輪、每輪384個突變體的分子克隆、誘導表達和質譜測試。在不使用先驗知識的前提下,BO-EVO通過模型-實驗閉環迭代,在1個月內實現底物選擇性指標近5倍的提升,達到文獻已報道最高水平。
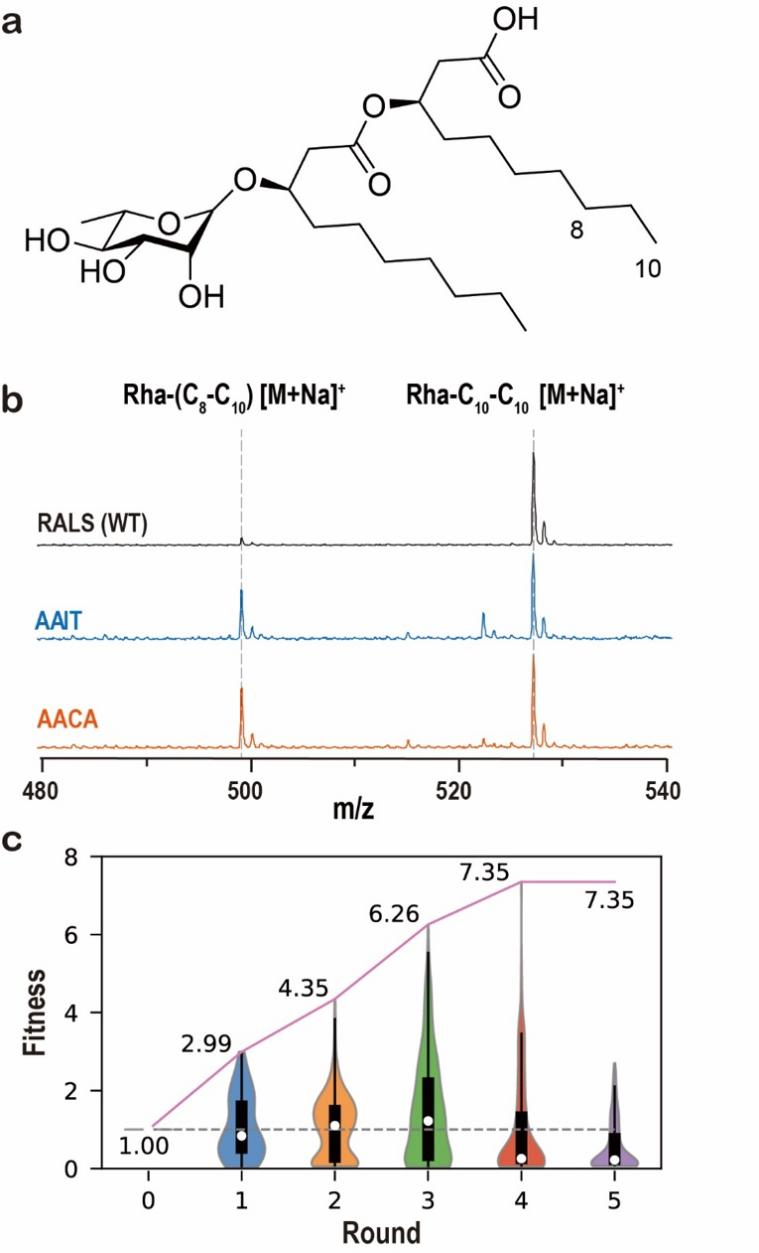
圖4. BO-EVO指導RhlA酶的4位點組合優化。a. 鼠李糖脂單體Rha-C10-C10分子結構;b. 鼠李糖脂單體的MALDI質譜檢測結果,包括野生型與典型變體;c. Rha-(C8-C10)的歸一化產量,野生型為1。
綜上,文章依托深圳合成生物大設施,開發了蛋白質適應度地形的高效探索算法,率先實現了基于機器學習模型與機器人實驗反饋迭代的自動化蛋白質工程改造。司同研究員、喬宇研究員和胡如云助理研究員為本文的共同通訊作者,胡如云助理研究員和博士生付立豪為本文共同第一作者。本研究得到國家重點研發計劃、國家自然基金及深圳合成生物學創新研究院等項目支持。
PI與課題組簡介:
司同,中科院深圳先進院合成生物學研究所研究員,博士生導師。國家重點研發計劃合成生物學項目首席科學家,國家高層次人才(青年),深圳合成生物研究重大科技基礎設施總工藝師。
課題組方向為自動化合成生物技術,包括機器學習指導蛋白工程、高通量質譜篩選等,用于開發微生物細胞工廠研究和生產燃料、化工品、藥物等重要分子,前期成果在Nat Commun, J Am Chem Soc, Angew Chem Int Ed, Chem Sci, Metab Eng等國際著名學術期刊發表論文50余篇,“谷歌學術”引用超過2400次。
實驗室主頁:
http://isynbio.siat.ac.cn/sitonglab/
附件下載:


