近日,中國科學院深圳先進技術研究院姜青山研究員、黃小羅高級工程師、中國農業科學院深圳農業基因組研究所戴俊彪研究員等聯合在國際學術期刊Small Methods (IF=12.4)上發表了題為An Effective DNA-Based File Storage System for Practical Archiving and Retrieval of Medical MRI Data的研究文章(圖1)。
文章中設計了一種名為"EDS"的DNA存儲方法,通過改進編碼模型、引入冗余核苷酸和設計索引技術,實現了醫學MRI數據的可靠歸檔和檢索。

圖1. 文章上線截圖
文章鏈接:https://doi.org/10.1002/smtd.202301585
醫學MRI數據是診斷、治療規劃和疾病監測等多種領域的重要工具。利用DNA存儲技術保存醫學MRI數據將有助于人類健康管理。通過DNA存儲,可以確保這些重要數據在數千年內安全保存并精確恢復,從而保證了這些重要數據的長期存儲。此外,DNA存儲還使得過去的健康數據更容易被未來的研究人員獲取,這對于縱向研究非常重要,因為它允許研究人員研究疾病的進展和治療效果。該工作提出了一種名為“EDS”的方法(圖2),通過三個關鍵組成部分實現了醫學MRI數據的歸檔。首先,研究團隊設計了一種新穎的分塊策略,解決了旋轉編碼導致的數據丟失問題。其次,提出了一種基于規則的四進制轉碼方法,滿足生化約束條件并確保可靠的數據映射。最后,設計了一種索引技術,簡化了隨機搜索和訪問過程。
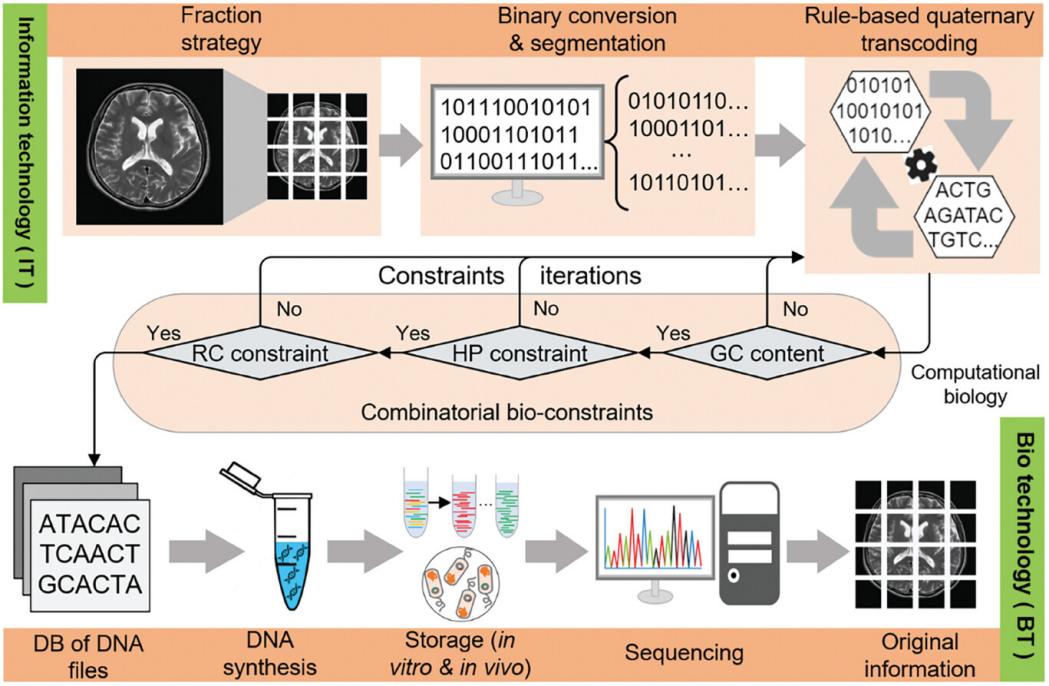
圖2. EDS方法流程圖
研究團隊提出了一種名為“DFS”的索引技術,克服了DNA文件存儲中高額外開銷的挑戰,旨在簡化DNA文件存儲的組織結構,實現靈活的隨機搜索、訪問和文件管理。
圖3中展示的正則標簽(RT,一個堿基對)有效地幫助搜索特定的分塊(子圖像),將其與其他分塊區分開來。圖像被分割為16個分塊,每個分塊被分配了不同的基因標簽,序列索引中設計了唯一的地址(4個堿基對),以精確定位和檢索所需的序列;此外,在隨機有效負載(平均長度為107個堿基對)之前添加了不同的標簽(DT,四個堿基對),用于區分每個分塊的解碼二進制數據,進而簡化了存儲數據的組織。
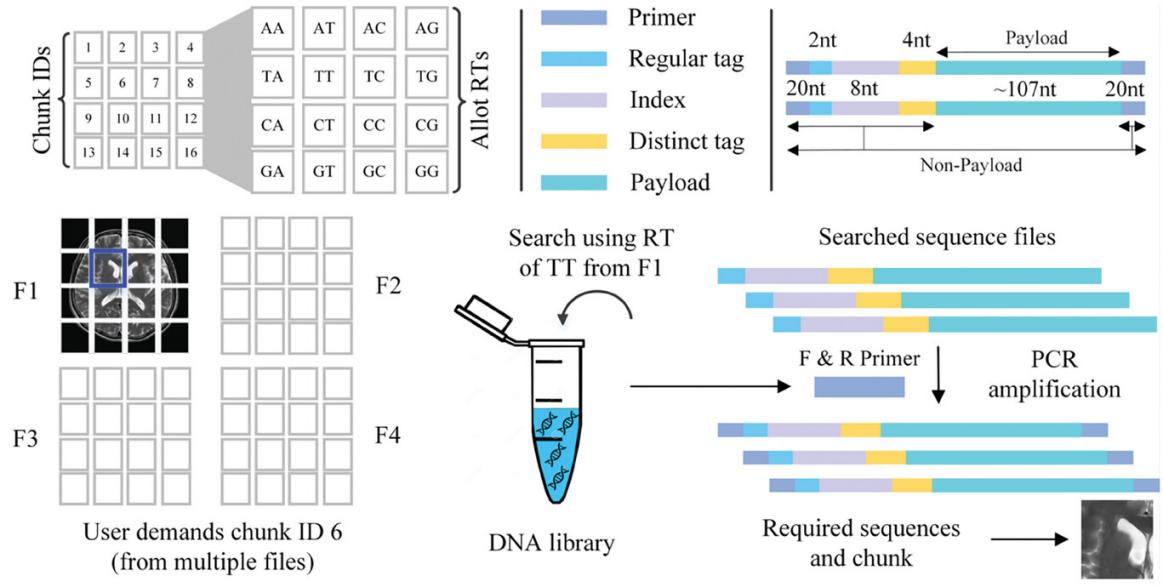
圖3. DFS技術示意圖
該工作在計算時間上也提出了新的方法,通過利用多進程技術優化DNA存儲編碼流程,將編碼任務分解成多個子任務,并分配多個CPU進行并行計算,提高編碼速率,實驗共測試了72GB的人體MRI數據,完成編碼僅需9個小時,編碼時間效率提升明顯,同時基于數據進行了預測,1TB量級的數據在120 h能完成。通過計算機模擬和生物合成實驗證實,EDS方法在醫學MRI數據存儲方面表現出色,并且具有更好的生化約束控制和較短的計算時間。為醫學MRI數據的DNA存儲開辟了新的途徑。
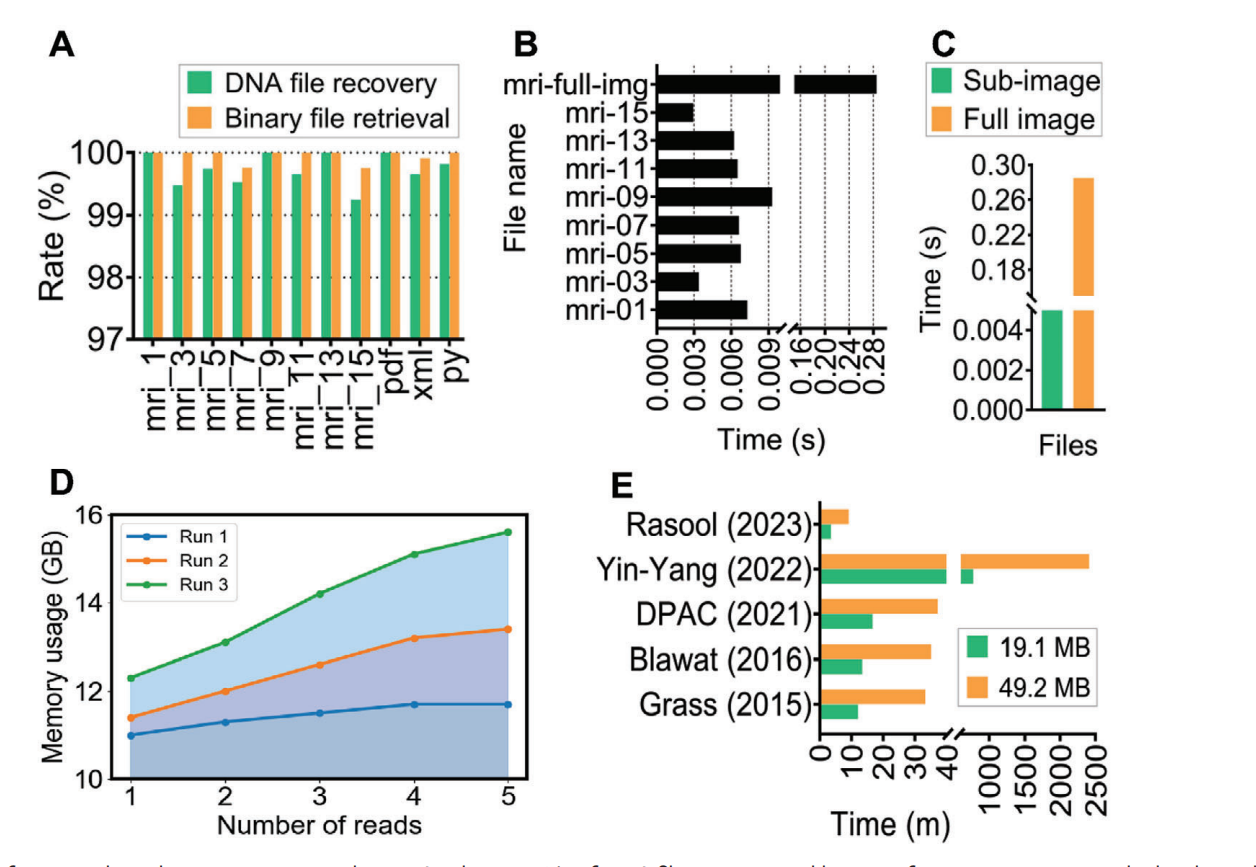
圖4. 體內合成驗證EDS 性能測試
這項研究成果為醫學領域的數據存儲和檢索提供了新的可能性,具有重要的實際應用前景。隨著進一步的研究和發展,DNA存儲技術有望成為醫學數據管理的重要工具,為醫學健康領域帶來更多的創新和進步。
中國科學院深圳先進技術研究院姜青山研究員、黃小羅研究員高級工程師,中國農業科學院深圳農業基因組研究所戴俊彪研究員為該文章的通訊作者,博士生Abdur Rasool、碩士生洪經緯為論文的共同第一作者。該研究獲得國家重點研發項目、深圳市科技項目等多個基金的資助。
附件下載:


